Texto: Antonio Tomás Palacios García (apalacios@labexcell.es), Eduardo Leiva Rebollar (comercial@labexcell.es) Laboratorio Excell Ibérica.
Brettanomyces y la uva
«¿Está presente Brettanomyces en las uvas?», «¿Las levaduras Brettanomyces que contaminan mi vino provienen de las uvas?» Estas dos preguntas, que son recurrentes en la época de la cosecha, parecen, a primera vista, muy similares. Pero cada uno tiene su propia respuesta.
A la primera, la respuesta ya no se debate. Numerosos estudios científicos lo han demostrado durante casi veinte años: sí que es posible detectar células de Brettanomyces bruxellensis en la superficie de las uvas en el viñedo. Para ello, son posibles dos opciones analíticas: o bien colocar las bayas en un medio de enriquecimiento que favorezca la detección de células de Brettanomyces mediante técnicas convencionales de microbiología, o bien utilizar una técnica de detección ultrasensible específica, como la PCR digital.
La respuesta a la segunda pregunta es más tendenciosa. Los estudios coinciden en que las cepas de Brettanomyces bruxellensis que están presentes en las uvas rara vez son las responsables de la contaminación por fenoles volátiles durante el envejecimiento del vino durante varios meses después, en la bodega. Por otro lado, las Brettanomyces presentes en las uvas pueden actuar metabólicamente, al igual que otras levaduras o bacterias presentes en la pruína de las uvas, durante las fases de vinificación. Esto depende de la dinámica de fermentación y de la competencia con las otras especies presentes en el medio.
Por lo tanto, es prudente analizar Brettanomyces en bayas de uva porque es perfectamente legítimo considerar que las uvas que tienen presencia de Brettanomyces en su superficie ofrecen una matriz más permisiva para su desarrollo posterior. Esto es cierto en la vinificación e incluso en la crianza si otras cepas de Brettanomyces bruxellensis (las domesticadas en bodega) intentan tomar el relevo a las de las uvas.
Seguimiento de las uvas en el laboratorio
En el laboratorio Excell Ibérica tenemos una retrospectiva inédita y exclusiva sobre el seguimiento de Brettanomyces en uva, habiendo seguido en este trabajo la trayectoria profesional de nuestro director general desde el inicio de su tesis doctoral durante la vendimia 2003 (Tabla 1).
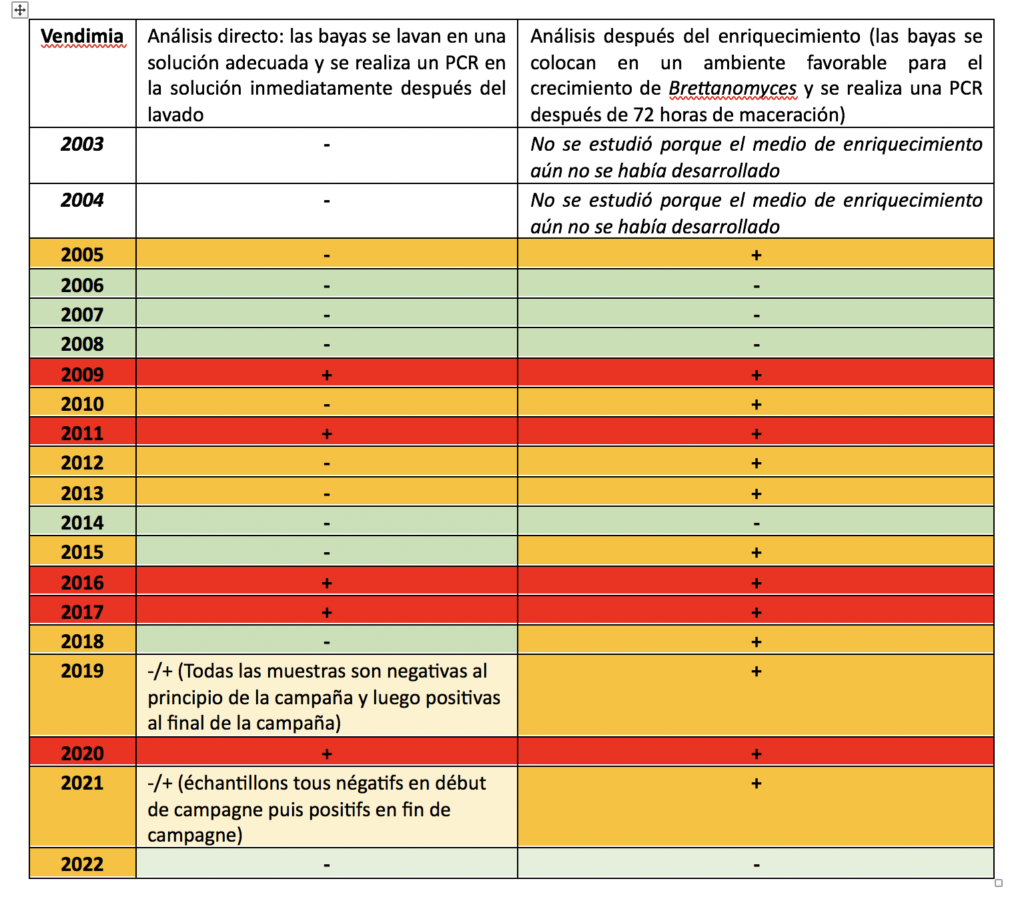
Desde 2022, el laboratorio también cuenta con una herramienta de biología molecular mucho más eficiente para cuantificar Brettanomyces bruxellensis: el PCR digital. Esta técnica es 100 veces más sensible que la PCR cuantitativa y 1000 veces más sensible que todas las técnicas de citometría. Permite detectar específicamente Brettanomyces bruxellensis sin enriquecimiento, lo que hace que el análisis sea totalmente cuantitativo (hasta ahora, el enriquecimiento hace perder la noción de la cantidad de células reales en la muestra original). La tecnología se aplicó por primera vez durante la cosecha de 2023.
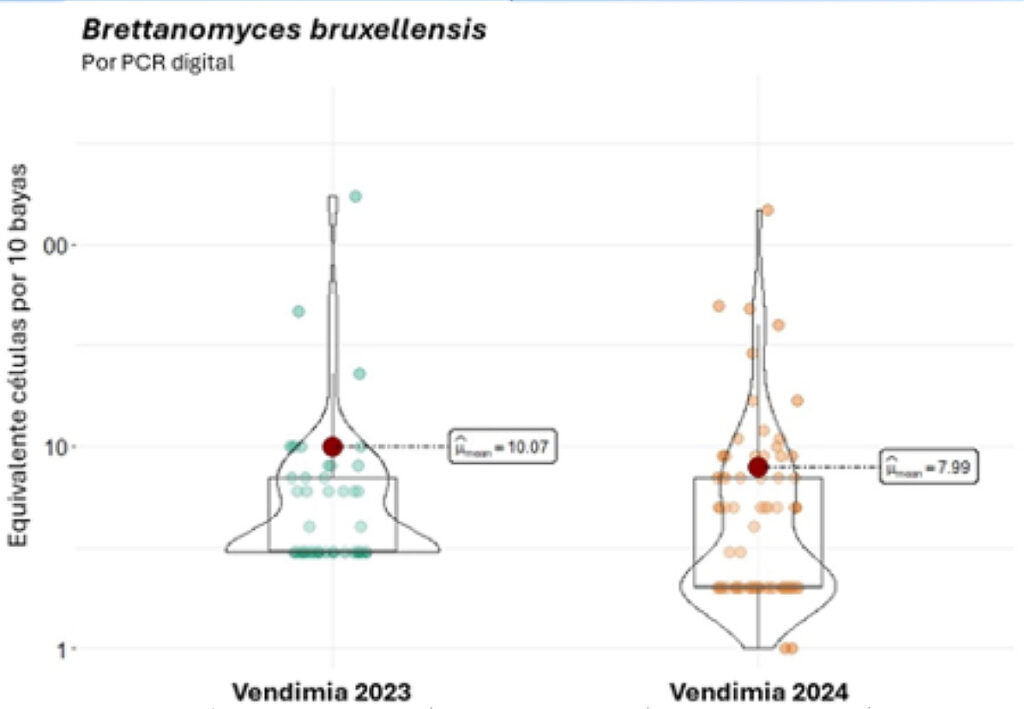
En 2023, de todas las uvas analizadas en el laboratorio por PCR digital en uvas (+ 200 muestras en total), la tasa de detección fue del 20% y la población media fue de unas 10 células por cada 10 bayas. En 2024, el 50% de las muestras procesadas hasta la fecha son positivas para Brettanomyces por PCR digital. Los datos de ambas añadas se refieren aproximadamente a la misma población media por baya de Brettanomyces: alrededor de 1 célula por baya. Sin embargo, la tasa de recurrencia en las uvas de 2024 es significativamente mayor que la de la cosecha anterior, lo que requiere un seguimiento lo antes posible de las poblaciones para evitar la contaminación temprana de fenoles volátiles.
Entonces, sobre el mosto y el vino procedente de la fermentación: cuidado con los métodos de análisis que no utilizan criterios genéticos de especificidad en su alcance.
Sobre la base de los resultados presentados anteriormente, el seguimiento de Brettanomyces en el mosto ha resultado particularmente útil este año, especialmente en el contexto de protocolos de vinificación particularmente arriesgados: maceración prefermentativa en frío, vinificación sin o después de poca sulfitación de la cosecha, fermentaciones con levaduras autóctonas, ausencia o estrujado débil, antecedentes de contaminación temprana de fenoles volátiles, etc. Para las cosechas más tardías, los riesgos de intrusión de Botrytis son lamentablemente probables y también se ha establecido que las uvas debilitadas por Botrytis toleran más a Brettanomyces que las bayas sanas, por lo que estos seguimientos tempranos serán necesarios en tales casos.
Pero la elección de la técnica analítica utilizada es fundamental.
Muchas especies diferentes están presentes en el mosto: desde Saccharomyces que comenzará a fermentar gradualmente, hasta levaduras no Saccharomyces. La más común en los mostos es Hanseniasporauvarum (p. ej. Kloeckera apiculata). Generalmente, esta especie tratará de mantenerse durante la fermentación alcohólica compitiendo con las otras especies presentes, incluyendo Saccharomyces cerevisiae. En cuanto a las otras levaduras no Saccharomyces (Metschikowia, Torulaspora, Pichia, Lachancea, etc.), que actualmente son objeto de numerosos estudios y ensayos, también contribuyen a una mezcla real de células.
En este entorno tan diverso, es esencial usar una técnica que utilice el criterio bioquímico más fino de especificidad, es decir, la genética, para buscar específicamente una especie como Brettanomyces. Todas las demás técnicas que puedan basarse en criterios fisiológicos y morfológicos, utilizando diferentes posibilidades de marcado, conducirán inevitablemente a una gran sobreestimación de la población real de Brettanomyces. Estas técnicas pueden conducir a una toma de decisiones errónea. Este es especialmente el caso de algunas metodologías de citometría que no utilizan el marcaje genético. La citometría es una tecnología muy potente, pero en un mosto en fermentación en el que conviven varios cientos de miles de levaduras, solo la citometría con sondas genéticas específicas da resultados satisfactorios. Cada año se realizan pruebas comparativas de seguimiento entre la PCR (siempre en base a una especificidad relacionada con los primers) y estas técnicas de citometría. Los resultados muestran una tendencia a la sobreestimación de las poblaciones en mostos por parte de estos últimos, muy significativa de forma precisa durante este año. De hecho, estas comparaciones son particularmente convincentes, ya que a veces se indican hasta 105/106 células por citometría y solo unas pocas docenas por PCR digital.
Más tarde, cuando el sistema ecológico se haya simplificado, cuando el etanol haya ejercido su efecto inhibidor sobre muchas especies y cuando sólo unas pocas células residuales de Saccharomyces puedan coexistir con Brettanomyces, la citometría que no utiliza sondas genéticas puede proporcionar resultados suficientes, pero en la fermentación del mosto y el vino recién termnado definitivamente no es el caso.
Conclusiones
En las uvas, mostos y durante la fermentación, es esencial mantener la cautela en la elección del método analítico para la cuantificación Brettanomyces bruxellensis. Es entonces aconsejables emplear métodos analíticos específicos dirigidos al ADN o que utilicen sondas de ARN.
Los resultados actualizados de 2024 muestran una frecuencia particularmente significativa de detección de Brettanomyces bruxellensis en uvas (1 de cada 2 muestras).
Con el fin de ser lo más preventivo posible, tiene sentido anticipar el seguimiento de las poblaciones de Brettanomyces desde el mosto y vinos recién terminados, pero para ello es necesario utilizar un método que sea a la vez específico y sensible. El PCR digital es la técnica más adecuada para este objetivo.
El análisis de ácidos fenólicos libres y esterificados, otro indicio de vulnerabilidad a Brettanomyces
Para producir fenoles volátiles, Brettanomyces utiliza ácidos fenólicos: ácido cumárico y ácido ferúlico. Estos ácidos fenólicos no están libres en las uvas, sino que están esterificados, el ácido cumárico en ácido cutárico y el ácido ferúlico en ácido fertárico. El análisis de este conjunto de precursores en las uvas y/o mostos y vinos recientes también puede revelar diferencias entre los diferentes lotes de cosecha. Al igual que con la detección temprana de Brettanomyces, no hay duda de que los lotes con concentraciones más altas de estos compuestos serán posteriormente más sensibles y deben ser monitoreados más de cerca.
Suscríbete gratis a nuestro boletín.¡Pincha aquí!












